CT Medical Image Classification
(0) 서론, 액션플랜
0. 데이터 구성
- dicom_dir : dicom 파일 폴더 (100 files)
- tiff_images: tiff 파일 폴더 (100 files)
- full_archive.npz: images, idx 어레이들의 집합(475 instances)
- overview.csv: 환자 ID, 환자의 특징과 dicom 파일명을 column으로 가지는 데이터 프레임(100 instances)
* npz?
변수를 저장하고 싶을 때가 있는데, 여러 개의 array 변수를 한 번에 저장하고 싶을때 사용하는 것이 npz
여러 개의 array를 담고 있기 때문에 하나의 array에 접근하기 위해서는 그 변수를 알아야 한다.
위의 full_archive.npz는 image와 idx라는 이름의 array 변수가 있었다. 따라서 아래와 같이 각각의 변수 타입을 프린트 하면
import numpy as np
x = np.load('full_archive.npz', allow_pickle=True)
print(type(x['image']))
print(type(x['idx']))아래와 같은 output이 나온다.

x['image'][:10]image 변수를 살펴보면 아래와 같다.

x['idx'][:10]idx를 뽑으면 이렇게 된다.
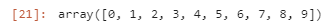
full_archive의 두 변수 image와 idx의 경우 475개의 개체가 있다.
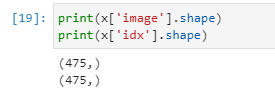
한편.. dicom_dir과 tff_images의 경우 100개의 개체뿐이다. overview.csv 에도 100개의 개체뿐이므로, full_archive는 이용하지 않도록 하겠다.
overview 파일은 아래와 같은 정보를 담고 있다.

- input: DICOM
- output: Contrast
1. DICOM 파일 열어보기
일단 dicom 파일 하나를 열어보자
https://hyelimkungkung.tistory.com/44
[의료이미지 분석]Introduction to medical image analysis
https://www.edwith.org/medical-20200327/joinLectures/30437 위 강조를 참조하여 작성하였습니다. 포스팅에 사용된 이미지 역시 위 강의의 슬라이드에서 가져왔습니다. @의료 이미지 분석이란? 의료 이미지..
hyelimkungkung.tistory.com
여기에 써 놨듯이 의료 이미지에는 영상 정보와 헤더 정보가 함께 있어야 한다. 한 dicom 파일에도 영상 정보와 헤더 정보가 함께 들어있다. 파이썬에서 dicom 파일을 열어보기 위해서 사용할 수 있는 것이 'pydicom' 라이브러리다.
이용해서 아래와 같이 하나의 무작위 파일을 열어서 읽어보았다.
!pip install pydicom
import pydicom
dicom = pydicom.read_file('dicom_dir/'+os.listdir('dicom_dir')[0])
그러면 아래와 같이 메타데이터에 대한 정보가 담겨있다.

정확하게 무엇에 대한 정보인지 알 수 없는 데이터이지만 조금 더 내려보면 우리가 해석할 수 있는 정보가 있다.

이미지 차원, width, height, pixel spacing(강의에서는 voxel spacing에 대해서 다루었지만 이미지가 2차원인 고로 pixel spacing인 것 같음)
흥미롭다!
2. DICOM 파일 array로 전환 및 시각화
data = dicom.pixel_array

위에서 본 image dimension과 shape가 정확히 같다.
따라서 data는 dicom의 영상 정보가 적절히 잘 전환되었다고 생각할 수 있다.
그러나 여기서 그쳐서는 안 된다고 한다.
window_center = -600
window_width = 1600
# CT image
dicom_path ='dicom_dir/'+os.listdir('dicom_dir')[0]
slice = pydicom.read_file(dicom_path)
s = int(slice.RescaleSlope)
b = int(slice.RescaleIntercept)
image = s * slice.pixel_array + b
plt.subplot(1,4,1)
plt.title('DICOM -> Array')
plt.imshow(slice.pixel_array,cmap='gray')
plt.subplot(1,4,2)
plt.title('Rescaled Array')
plt.imshow(image, cmap = 'gray')
# apply_voi_lut( )
slice.WindowCenter = window_center
slice.WindowWidth = window_width
image2 = apply_voi_lut(image, slice)
plt.subplot(1,4,3)
plt.title('apply_voi_lut( )')
plt.imshow(image2, cmap = 'gray')
# normalization
image3 = np.clip(image, window_center - (window_width / 2), window_center + (window_width / 2))
plt.subplot(1,4,4)
plt.title('normalize')
plt.imshow(image3, cmap = 'gray')
plt.show()

총 3가지의 변화를 주었는데 아래와 같다.
(1) rescaleslope, rescaleintercept 반영
(2) apply_voi_lut
(3) normalize
(1) rescaleslope, rescaleintercept 반영
우선 resclaeslope과 rescaleintercept를 반영했을 때 그림상으로는 무엇이 크게 달라지는지는 잘 모르겠다.
하지만 값에서는 확연히 차이를 보인다.
 |
 |
왼쪽이 rescaleslope와 intercepty를 반영하지 않았을 때의 array 값, 그리고 오른쪽이 반영했을 때의 값이다.
모두 음수로 바뀐 것을 알 수 있다. 그렇다면 왜 그렇게 됐는가? 그 원리가 궁금하면 아래의 블로그를 참조하도록 하자.
https://jayeon8282.tistory.com/2
(2) apply_voi_lut
영상을 통해서 어떤 차이를 주었는지 한 번에 확인할 수 있다. 이미지의 밝기가 변해서 우리가 쉽게 이해할 수 있는 구조로 변하였다.
(3) normalize
솔직히 말하면 사진이 작아서 어떤 변화가 일어났는지 잘 파악을 할 수 없다.
하지만 수식적으로는 그 차이가 무엇인지 알 수 있을 것이다.
우리가 임의로 정한 Window_center 와 window_width 값을 넘는 것들은 모두 컷해서 우리가 지정한 값들이 되게끔 하는 장치이다.
3. 위에 기반하여 dicom을 array로 전환하는 함수 생성
def dicom_2_array(path):
window_center = -600
window_width = 1600
slice = pydicom.read_file(path)
s = int(slice.RescaleSlope)
b = int(slice.RescaleIntercept)
image = s * slice.pixel_array + b
# apply_voi_lut( )
slice.WindowCenter = window_center
slice.WindowWidth = window_width
image2 = apply_voi_lut(image, slice)
return image2
4. contrast 사용 여부가 다른 dicom 파일 시각화
## contrast 가 True인 이미지와 False인 이미지 섞어서 subset 생성
df = pd.read_csv('overview.csv')
df_contrast = df.loc[df.Contrast == True,:][:8]
df_none = df.loc[df.Contrast == False,:][:8]
df_subset =pd.concat([df_contrast, df_none]).reset_index(drop=True)
## subset의 시각화
f, ax = plt.subplots(4,4, figsize=(16,20))
for i, data in enumerate(df_subset.values):
path = 'dicom_dir/' + data[-1]
image = dicom_2_array(path)
ax[i//4, i%4].imshow(image, cmap='gray')
ax[i//4, i%4].set_title(f'Age: {data[1]}\nContrast: {data[2]}')

시각화를 했을 때 어떤 차이가 있는 것 같은가?
솔직히 말하면 나는 아직 잘 모르겠다.
5. 데이터 프레임에 pixe_array column 추가하기
나중에 작업하기 용이하도록 pixel_array를 그냥 column으로 새로 만들어준다.
pixel_array = []
for path in df.dicom_name:
dicom_path = 'dicom_dir/' + path
array = dicom_2_array(dicom_path)
pixel_array.append(array)
df['pixel_array'] = pixel_array
오늘은 쉽게 끝낼 수 있었다. 다행이다. ㅠㅠ
나도 프로젝트 할 때마다 표지를 만들어서 게시할 수 있으면 참 좋을 것 같다.
좀 깔끔하고 기분이도 좋고
[reference]
- https://www.kaggle.com/raddar/convert-dicom-to-np-array-the-correct-way
'딥러닝 > 프로젝트' 카테고리의 다른 글
| [과제] 당뇨병 환자 클러스터링 (0) | 2021.09.29 |
|---|---|
| [Kaggle] CT Medical Image - (2) DataLoader 만들기 (0) | 2021.09.27 |
| [Kaggle] CT Medical Image - (0) 서론, 액션 플랜 (0) | 2021.09.25 |
| [개인]R-CNN 을 이용한 BCCD type 분류 -(3)linear svm, (4)결론 (0) | 2021.06.29 |
| [개인]R-CNN 을 이용한 BCCD type 구분 -(2) fine tuning (0) | 2021.06.29 |



댓글